Diffusion models for protein design
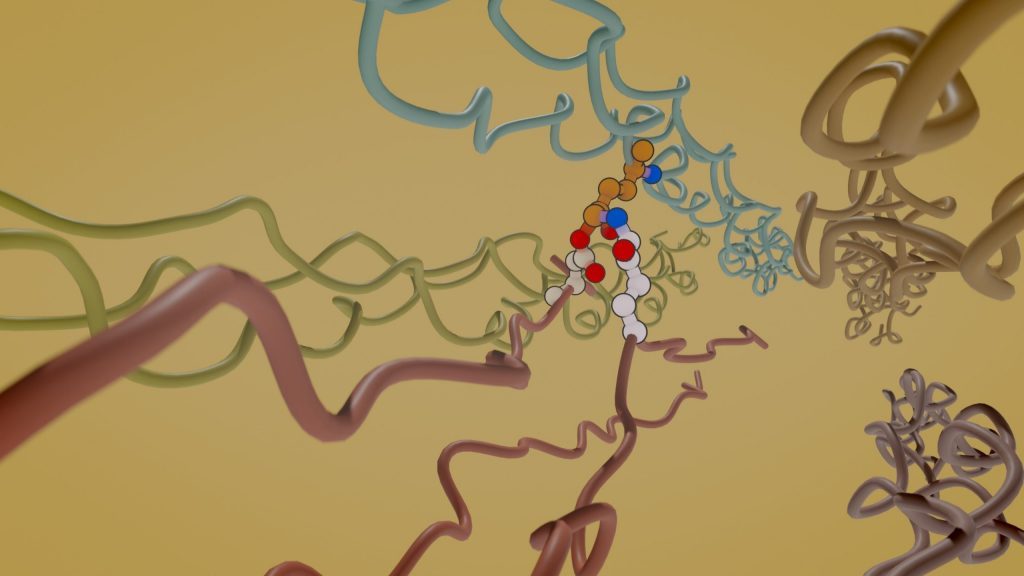
Generatives Deep Learning hat das Proteindesign verändert und ermöglicht die Entwicklung neuer Proteinstrukturen, Interaktionen und Funktionen mit hoher Präzision, durch generative Modelle wie Diffusionsmodelle und Flow Matching. Geometrisches Deep Learning spielt eine entscheidende Rolle, da es die Modellierung von Symmetrien ermöglicht, um die Aussagekraft und Effizienz des Modells zu optimieren.
In diesem Projekt zielen wir darauf ab, aktuelle generative Modelle für das Proteindesign zu verbessern, indem wir die Art und Weise, wie Äquivarianz in sie eingebaut wird, überdenken und diese Erkenntnisse in den generativen Prozess einbeziehen. Wir befassen uns speziell mit dem Design eines Proteins mit einer dreiwertigen Querverbindung: einer komplexen chemischen Bindung zwischen drei Aminosäuren. Eine solche Struktur wurde noch nicht erfolgreich synthetisiert, es ist aber bekannt, dass sie in Kollagen vorkommt. Diese Herausforderung nehmen wir an und wollen damit sowohl die Diffusionsmodelle verbessern als auch unser Verständnis dieses komplizierten biochemischen Prozesses vertiefen.
Beteiligte:
Frauke Gräter (MBM)
Jan Stühmer (MLI)
Mitglieder:
Leif Seute
Vsevolod Viliuga
Simon Wagner
Nicolas Wolf
Publikationen:
Seute et al.: Grappa — A Machine Learned Molecular Mechanics Force Field
https://arxiv.org/abs/2404.00050
Wagner S, Seute L, Viliuga V, Wolf N, Gräter F, Stühmer J: Generating Highly Designable Proteins with Geometric Algebra Flow Matching.
The Thirty-eighth Annual Conference on Neural Information Processing Systems (NeurIPS) 2024 (accepted).