CompIF: Effiziente Berechnung der intermolekularen Kräfte für biomolekulare Simulationen
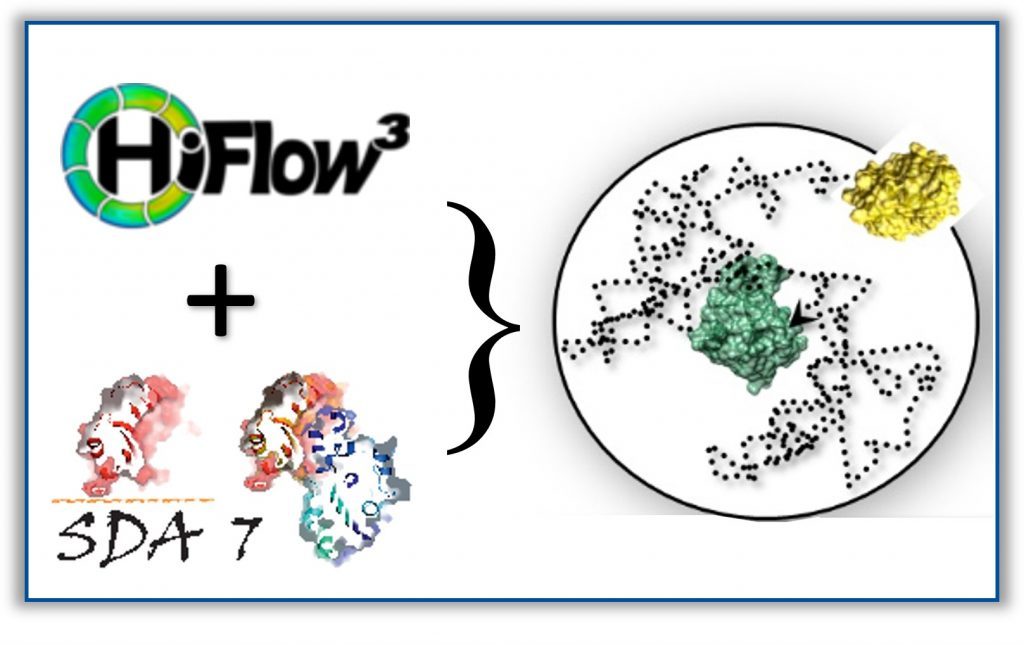
Die Struktur, Dynamik und Wechselwirkungen von Biomolekülen können mit Molekülsimulationen im Detail erforscht werden. Durch wiederholtes Lösen der Bewegungsgleichungen in kurzen Zeitabständen werden Trajektorien (oder Filme) der zeitlichen Entwicklung molekularer Systeme erzeugt. Moleküle bewegen sich aufgrund der auf sie wirkenden Kräfte. Deshalb ist die genaue Berechnung der Kräfte von entscheidender Bedeutung, sie ist aber auch generell der rechenintensivste Teil einer Molekularsimulation. Daher sind effiziente Algorithmen und Näherungen erforderlich, um biomolekulare Systeme zu simulieren.
In diesem HITS Lab-Projekt verbessern wir die Berechnung intermolekularer Kräfte in Brownschen Dynamiksimulationen der Proteindiffusion, die mit dem in der MCM-Gruppe entwickelten SDA-Softwarepaket durchgeführt werden. Zu diesem Zweck werden schnelle, hardwarenahe Solver zur Berechnung von Kräften in das in der DMQ-Gruppe entwickelte HiFlow3-Softwarepaket für finite Elemente implementiert. Wir schaffen Schnittstellen zwischen den beiden Softwarepaketen und setzen Methoden maschinellen Lernens ein, um Simulationstrajektorien effizient zu generieren. Die Ergebnisse werden den Anwendungsbereich der Brownschen Dynamiksimulationen für die Untersuchung von Proteinen in verdünnten und makromolekular gefüllten Umgebungen erweitern.
Beteiligte:
Rebecca Wade (MCM)
Vincent Heuveline (DMQ)
Mitglied:
Riccardo Beccaria