Projekte
Unser Hauptziel ist die Entwicklung und Anwendung computergestützter Methoden zur Modellierung, Simulation und Vorhersage der Interaktion von Biomolekülen, wobei der Fokus auf der Interaktion von Proteinen liegt. Dazu nutzen wir dreidimensionale makromolekulare Strukturen und kombinieren Ansätze, die auf physikochemischen Prinzipien beruhen, mit Chemo- und Bioinformatik. Einige Projekte werden auf dieser Seite näher erläutert (Die Inhalte sind nur auf Englisch verfügbar).
CompIF: Effiziente Berechnung der intermolekularen Kräfte für biomolekulare Simulationen
Die Struktur, Dynamik und Wechselwirkungen von Biomolekülen können mit Molekülsimulationen im Detail erforscht werden. Durch wiederholtes Lösen …
Mehr erfahren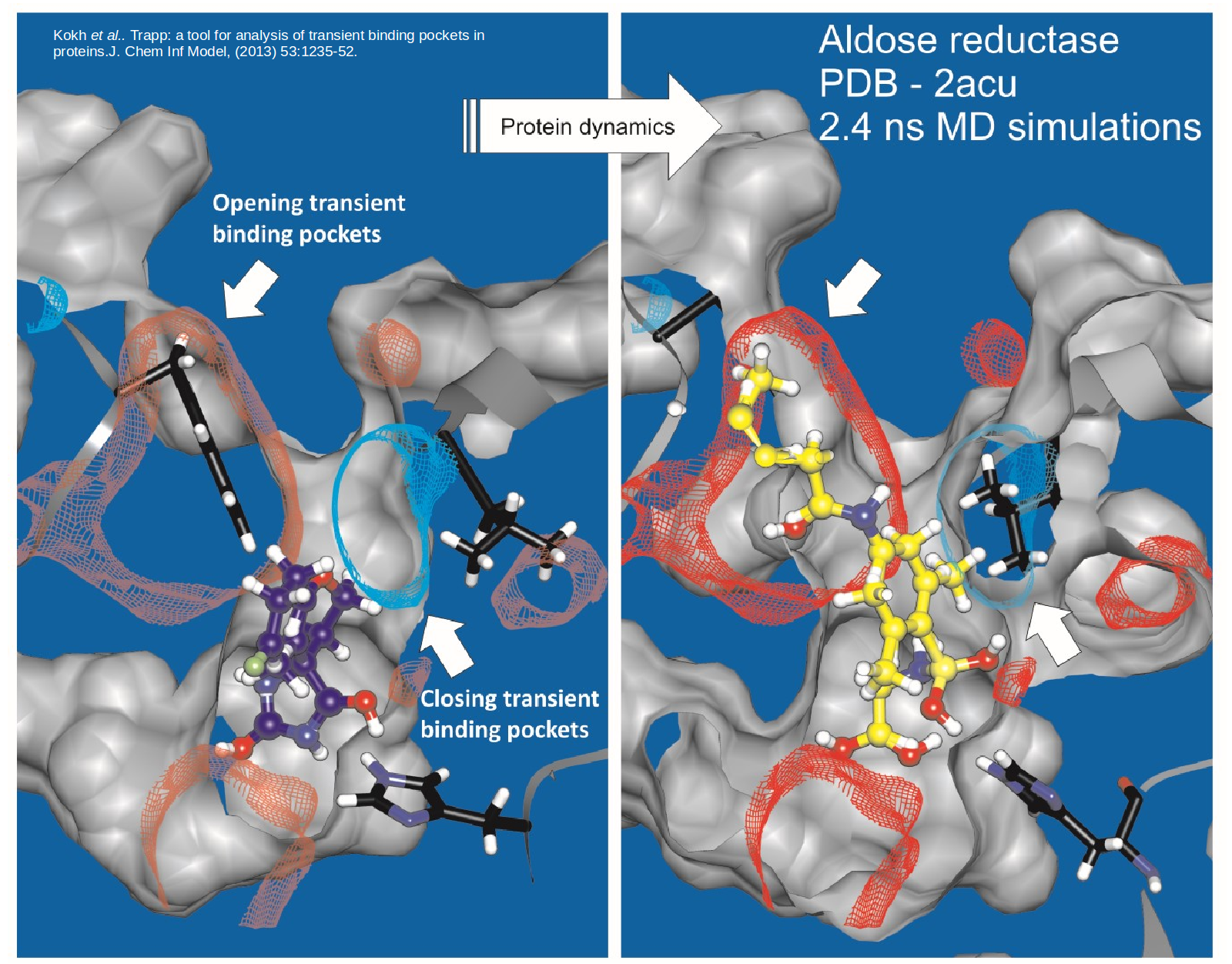
Structure-based drug discovery
Proteins are dynamic and constantly changing their shape. This flexibility not only presents a challenge to to structure-based drug design approaches …
Mehr erfahren
Macromolecular interactions and diffusional association
We are developing methods to predict protein-protein interactions and how proteins bind to surfaces. These methods rely on Brownian dynamics simulation …
Mehr erfahren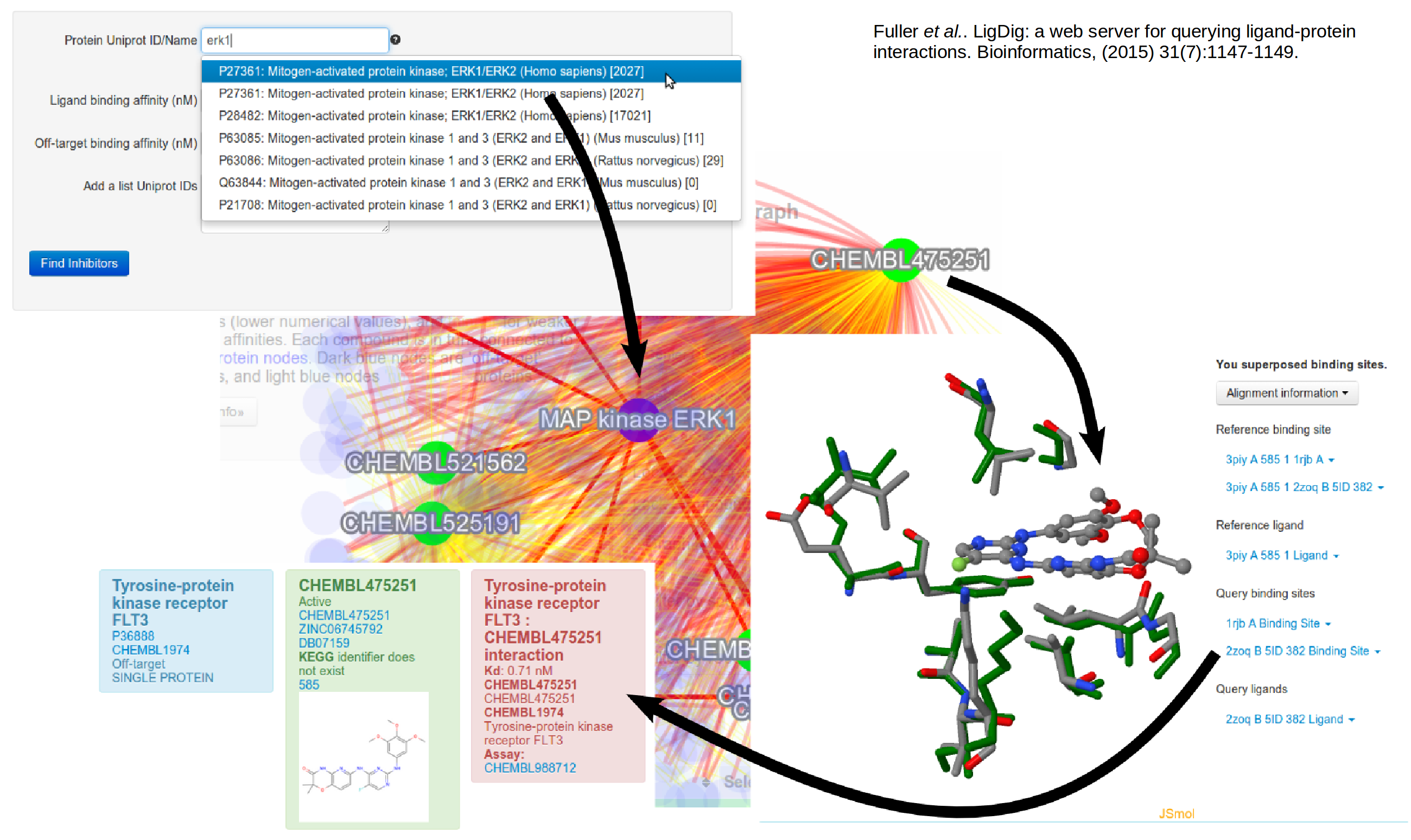
Protein structures in systems biology
We are working on developing approaches to bridge between protein structures and biochemical networks, from the molecular to the cellular level….
Mehr erfahrenGalerie

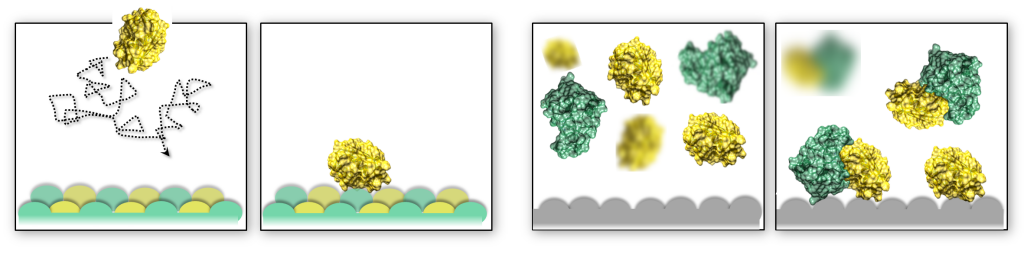
The shape of macromolecular crowders affects protein diffusion 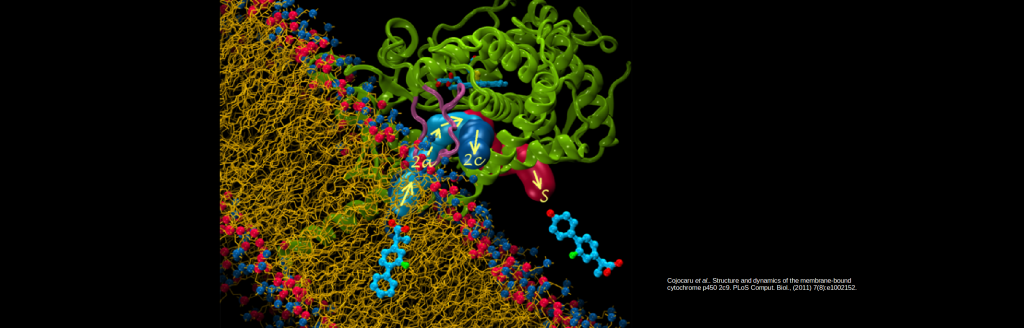
Simulations identify routes in and out of cytochrome P450 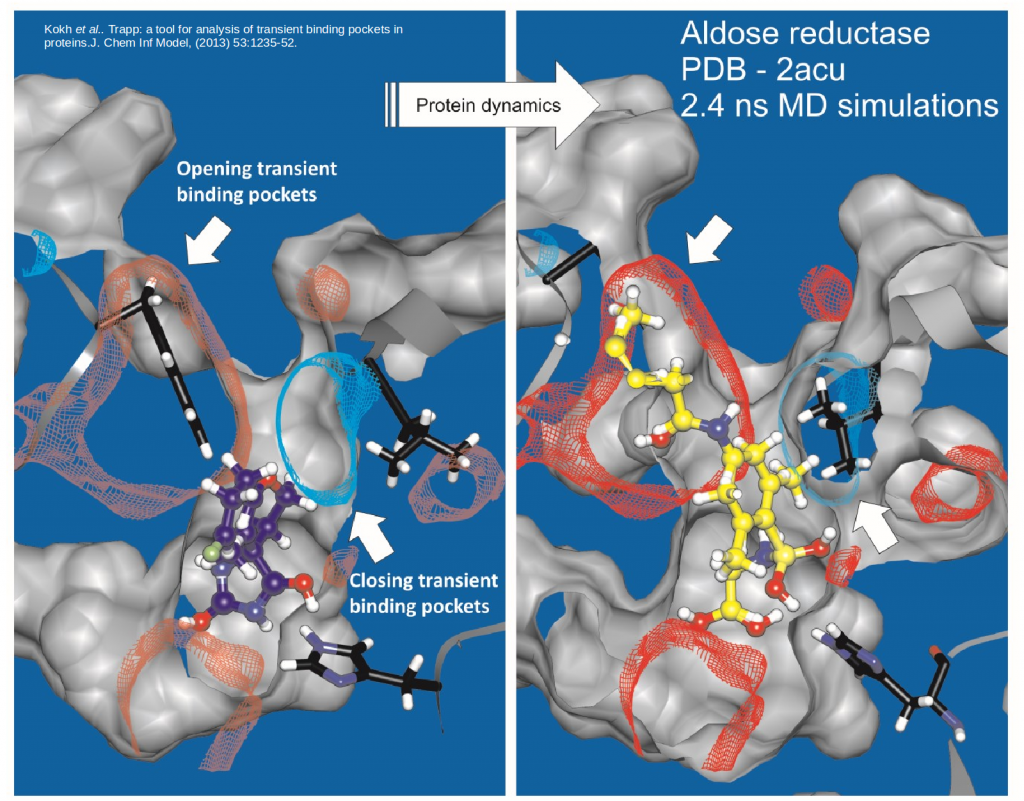
TRAPP helps to find transient pockets in proteins 
Simulations show how sickle cell hemoglobin starts to oligomerize 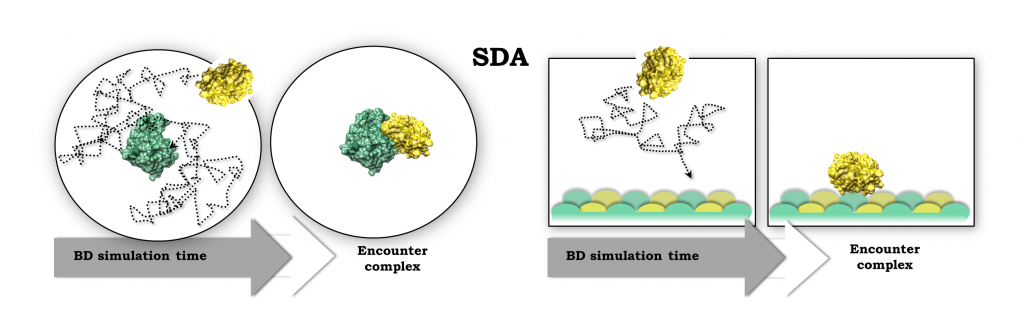
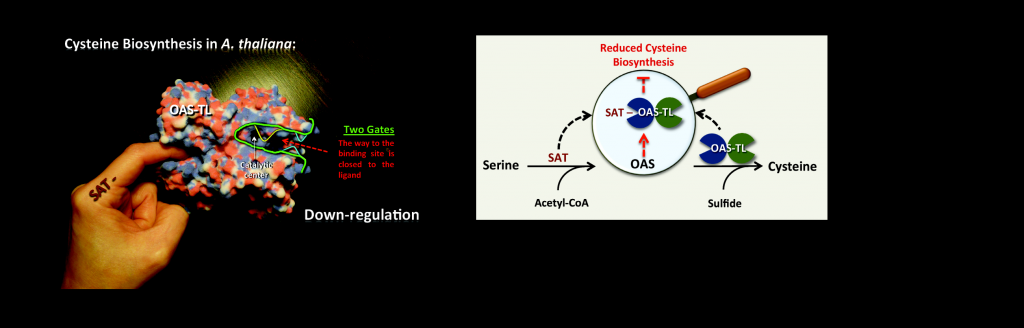
Allosterically gated enzyme dynamics regulate cysteine biosynthesis 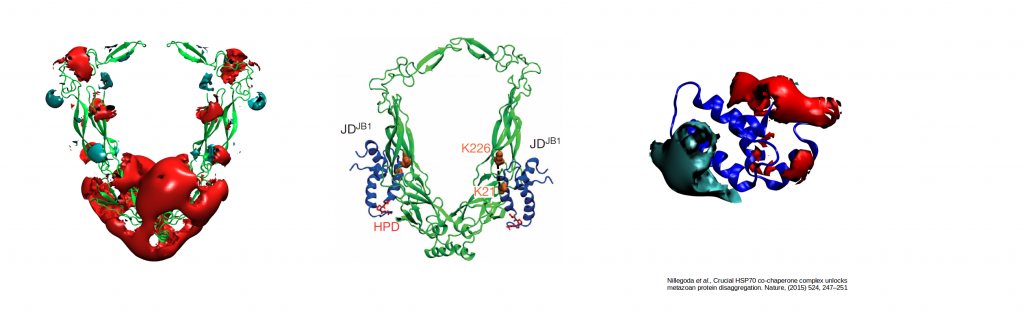
Electrostatically-guided Brownian dynamics docking of two co-chaperone J-proteins 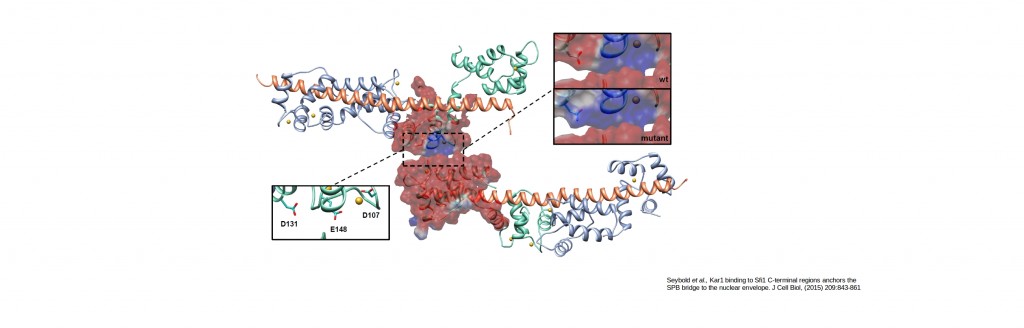
Free energy calculations for proteins stabilizing the spindle pole body bridge
